Resumen
Desde comienzos de la década del 2000, se han desarrollado sistemas de vigilancia basada en eventos (EBS) con el fin de recopilar información sobre epidemias publicada en los medios en línea (por ejemplo, Promed-mail desde 1994, HealthMap desde 2006, PADI-web desde 2016, EIOS desde 2017) para una serie de enfermedades y síndromes en humanos y animales. Esos EBS representan sistemas de alerta de eventos sanitarios que ayudan a los epidemiólogos a realizar una vigilancia sanitaria precoz, incluso en las zonas en las que los sistemas de vigilancia oficiales no están lo suficientemente presentes. Algunos de esos sistemas EBS, como PADI-web, integran métodos de inteligencia artificial (IA) y procesamiento del lenguaje natural (PLN) para mejorar los procesos de clasificación automática de documentos o identificar eventos epidemiológicos en los artículos. Este artículo ofrecerá ejemplos del uso cotidiano de los sistemas EBS en Francia.
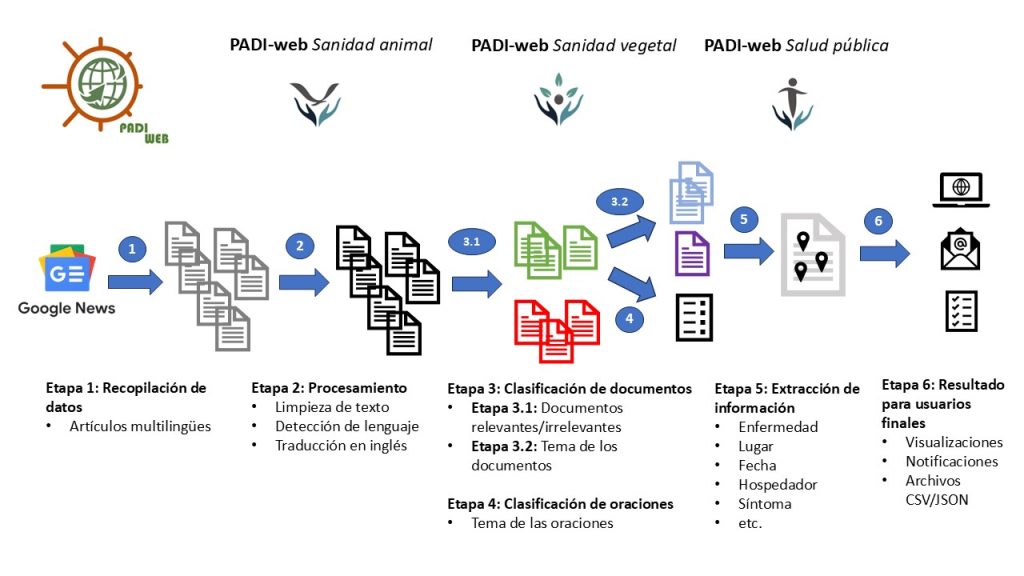
Figura 1. Proceso de procesamientos de PADI-web
La inteligencia epidemiológica es una disciplina que adquiere cada vez más importancia para la identificación mundial y precoz de enfermedades animales infecciosas y emergentes que pueden poner en peligro la sanidad del ganado a nivel nacional, la salud pública (zoonosis) y la conservación de las especies (razas con pocos ejemplares, fauna silvestre). Su finalidad es la identificación precoz, el análisis y el seguimiento de signos relativos a los peligros sanitarios que representen una amenaza para la sanidad animal. Se basa en la vigilancia continua de distintos tipos de signos de origen oficial y no oficial (medios de comunicaciones, redes sociales, etc.).
Una de las misiones de la Organización Mundial de Sanidad Animal (OMSA) es mejorar la detección precoz y la difusión de información relativa a la aparición de enfermedades animales en el mundo. Los Miembros de la OMSA transmiten información sanitaria oficial reglamentaria, estructurada y normalizada, relativa a las enfermedades animales a través de la base de datos WAHIS (World Animal Health Information System). Los expertos en inteligencia epidemiológica pueden analizar esos datos siguiendo una rutina predefinida que permite generar indicadores epidemiológicos y realizar análisis periódicos de situación. Sin embargo, múltiples factores pueden afectar a los datos oficiales o retrasar su notificación [1].
Un Nuevo Enfoque: Eventos sanitarios extraídos de la Web
Desde la década del 2000, una nueva generación de sistemas de vigilancia sanitaria completa el sistema que ya existe, identificando los eventos sanitarios obtenidos en internet y en otros medios electrónicos. Se trata de la vigilancia basada en eventos, o EBS, por sus siglas en inglés [2]. En el sector de la salud pública, incluida la sanidad animal específicamente, han surgido numerosas interfaces, como Promed (en 1994), HealthMap (en 2006), PADI-web (en 2016) y EIOS (en 2017), las cuales permiten identificar, extraer, clasificar visualizar los eventos sanitarios a partir de información textual y no estructurada.
Algunos de esos sistemas EBS, como PADI-web (Platform for Automated extraction of animal Disease Information from the Web) [3,4] integran métodos de inteligencia artificial (IA) [5] y procesamiento del lenguaje natural (PLN) para mejorar los procesos de clasificación automática de noticias (ver etapas 3 y 4 de la Figura 1) o identificar la información epidemiológica en los artículos (ver etapa 5 de la Figura 1). Se ofrecen interfaces específicas para la búsqueda de información (Figura 2) y la visualización de eventos epidemiológicos (Figura 3).
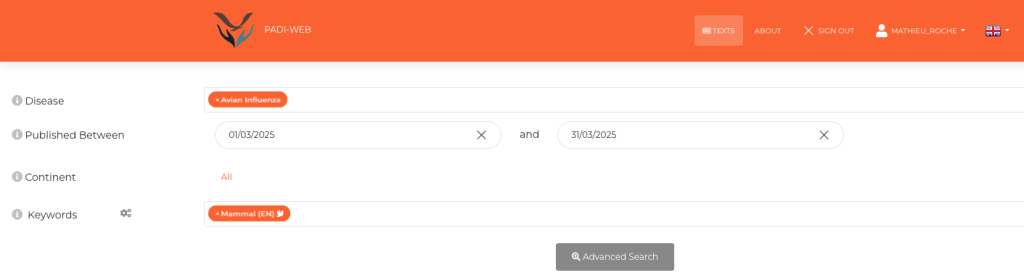
Figura 2. Interfaz de investigación de PADI-web
Para implementar y adaptar los enfoques de la IA al servicio de la vigilancia en sanidad animal, PADI-web utiliza datos anotados manualmente con expertos. Los datos sin procesar, recogidos y anotados para la sanidad animal, representan un material valioso que permite aprender de modelos específicos, utilizando, por ejemplo, métodos de aprendizaje automático. Asimismo, con los enfoques de la IA basados en modelos de lenguaje generales (por ejemplo, BERT – Bidirectional Encoder Representations from Transformers [6]) o más especializados (por ejemplo, BioBERT para el ámbito de la biomedicina [7], AgriBERT para la agricultura [8]), es posible adaptar los modelos a los ámbitos específicos y a las tareas por realizar. BERT es un modelo de lenguaje basado en una arquitectura específica con un mecanismo de atención que permite «comprender» las relaciones entre las palabras de una misma frase.
En el marco del proyecto MOOD (MOnitoring Outbreaks for Disease surveillance in a data science context), coordinado por Elena Arsevska, el CIRAD (Centro francés de Cooperación Internacional en Investigación Agrícola para el Desarrollo) y el INRAE (Instituto Nacional francés de Investigación para la Agricultura, la Alimentación y el Medio Ambiente) llevaron a cabo trabajos pluridisciplinarios para implementar e integrar modelos de IA en las herramientas de vigilancia epidemiológica. Se crearon corpus (conjunto de noticias) específicos a través de sesiones de anotación de datos con expertos en sanidad animal y la organización de «hackathons» (trabajo colectivo destinado a la realización de tareas específicas) con el fin de construir modelos de aprendizaje automático tradicionales (Support Vector Machine, Random Forest, etc.) y/o adaptar modelos de lenguaje. Dichas sesiones de anotación pueden basarse en métodos específicos, como el método Delphi (varias rondas de anotación para llegar a un consenso) y cálculos de acuerdo entre anotadores para consolidar guías de anotación y mejorar la calidad de los datos producidos que utilizan los algoritmos de IA.
Cada artículo del corpus utilizado se anota, determinando si una noticia es pertinente o no. Se considera que una noticia es pertinente si el texto describe un brote epidémico nuevo, sospechoso o desconocido. Para construir un modelo de extracción de información a partir de textos pertinentes, también debe extraerse la información epidemiológica (por ejemplo, enfermedades, hospedadores, lugares, fechas, etc.) del contenido textual. En general, los modelos de IA aprendidos y adaptados aplicando ajustes finos a partir de los datos anotados se integran actualmente en el funcionamiento del sistema PADI-web.
A través de estos enfoques de la IA es posible determinar automáticamente (i) si los documentos recopilados automáticamente en la red mediante un proceso de análisis e indexación son pertinentes, (ii) cuáles son los temas que transmiten y (iii) cuáles son las informaciones epidemiológicas (enfermedades, lugares de aparición de brotes, hospedadores, síntomas, número de casos, etc.) en los textos. Esta integración tiene en consideración los aspectos cualitativos de los resultados, así como la sencillez de los algoritmos empleados, lo cual constituye otro desafío decisivo de los métodos de IA que se desarrollan en la actualidad.
Hoy en día, se analiza una multitud de desafíos diferentes, por ejemplo, para identificar signos insignificantes, eventos idénticos o nuevos, etc. En este contexto, los modelos de lenguaje y los grandes modelos de lenguaje (técnicas de modelos generativos basados en ChatGPT, entre otros) pueden resultar particularmente eficaces, como lo demuestran los trabajos en colaboración entre investigadores de Strathmore University (Kenia) y el CIRAD [9].
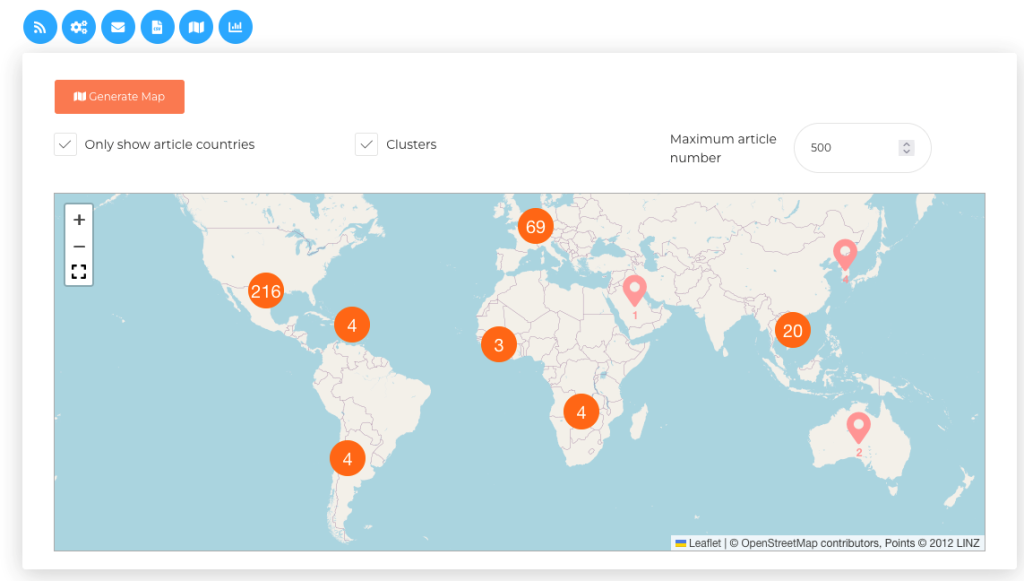
Figura 3. Interfaz de visualización espacial de eventos epidemiológicos de PADI-web
El CIRAD y el INRAE también han realizado trabajos sobre la vigilancia sindrómica en sanidad animal, la cual representa otro desafío para identificar nuevas enfermedades que pueden aparecer en nuevos territorios o en hospedadores nuevos. En este contexto, se llevó a cabo un análisis retrospectivo de enfermedad emergente, como la detección del virus de la influenza aviar de alta patogenicidad (IAAP) en especies hospedadoras inhabituales. Se identificaron siete casos de estudio de IAAP en mamíferos en la base de datos WAHIS, para los cuales se validaron manualmente los artículos recopilados por PADI-web [10]. Se evaluaron varias estrategias de clasificación de los lugares con el fin de privilegiar la sensibilidad o la especificidad del sistema de vigilancia.
Paralelamente a este trabajo de investigación realizado en el marco del proyecto MOOD, se realizaron sesiones de formación con usuarios finales así como fichas educativas [11] con el fin de facilitar el uso de la herramienta.
La unidad de inteligencia epidemiológica de la plataforma francesa de vigilancia epidemiológica de enfermedades animales (plataforma ESA) [12] utiliza diariamente estos sistemas EBS. Asimismo, uno de los desafíos actuales es fusionar esta variedad de información epidemiológica procedente de diversas fuentes. En este contexto, se ha desarrollado una nueva herramienta llamada MUST (Multi-Source Surveillance Tool) para establecer la correspondencia entre diferentes fuentes de datos sanitarios [10]. La primera parte de MUST está dedicada a la vigilancia de brotes de IAAP en mamíferos.
La herramienta recopila, filtra y mapea eventos de IAAP en mamíferos a partir de notificaciones oficiales extraídas de la base de datos WAHIS, datos oficiales de bases de datos en línea administradas por autoridades sanitarias (USDA para Estados Unidos de América y APHA para el Reino Unido) y eventos de diferentes EBS (Promed-mail y PADI-web). La herramienta se encuentra actualmente en fase de prueba para evaluar las diferentes estrategias de fusión según criterios espaciales, temporales y la información sobre los hospedadores implicados en los eventos epidemiológicos. Esta experiencia permitirá proponer enfoques a aplicar para los futuros usuarios de MUST.
Los enfoques de la IA pueden resultar especialmente eficaces para abordar los problemas relacionados con la vigilancia en sanidad animal. Sin embargo, estos trabajos requieren conocimientos especializados para anotar, validar los resultados de los algoritmos, ajustar los parámetros y operativizar las herramientas, lo que demanda la implementación de investigaciones multidisciplinarias.
Traducido del original en francés.
Copyright de la imagen principal: akinbostanci
Referencias
[1] Lin SY, Beltran-Alcrudo D, Awada L, Hamilton-West C, Lavarello Schettini A, Cáceres P, et al. Analysing WAHIS Animal Health Immediate Notifications to Understand Global Reporting Trends and Measure Early Warning Capacities (2005–2021). Transbound. Emerg. Dis. 2023. https://doi.org/10.1155/2023/6666672
[2] Paquet C, Coulombier D, Kaiser R, Ciotti M. Epidemic intelligence: a new framework for strengthening disease surveillance in Europe. Euro Surveill. 2026:11(12);5‑6. https://doi.org/10.2807/esm.11.12.00665-en
[3] Arsevska E, Valentin S, Rabatel J, de Goër de Hervé J, Falala S, Lancelot R, et al. Web monitoring of emerging animal infectious diseases integrated in the French Animal Health Epidemic Intelligence System. PLoS One. 2018:13(8);25. https://doi.org/10.1371/journal.pone.0199960
[4] Valentin S, Arsevska E, Rabatel J, Falala S, Mercier A, Lancelot R, et al. PADI-web 3.0: A new framework for extracting and disseminating fine-grained information from the news for animal disease surveillance. One Health. 2021:13. https://doi.org/10.1016/j.onehlt.2021.100357
[5] Sobkowich KE. Demystifying artificial intelligence for veterinary professionals: practical applications and future potential. Am. J. Vet. Res. 2025:86. https://doi.org/10.2460/ajvr.24.09.0275
[6] Devlin J, Chang MW, Lee K, Toutanova K. BERT: Pre-training of Deep Bidirectional Transformers for Language Understanding. Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. 2019:1;4171-86. https://doi.org/10.18653/v1/N19-1423
[7] Lee J, Yoon W, Kim S, Kim D, Kim S, Ho So C, et al. BioBERT: a pre-trained biomedical language representation model for biomedical text. Bioinformatics. 2020:36(4);1234-40. https://doi.org/10.1093/bioinformatics/btz682
[8] Rezayi S, Liu Z, Wu Z, Dhakal C, Ge B, Zhen C, et al. AgriBERT: Knowledge-Infused Agricultural Language Models for Matching Food and Nutrition. Proceedings of the Thirty-First International Joint Conference on Artificial Intelligence – AI for Good. 2022;5150-56. https://doi.org/10.24963/ijcai.2022/715
[9] Menya E, Roche M, Interdonato R, Owuor D. EpidGPT: A combined strategy to discriminate between redundant and new information for epidemiological surveillance systems. In: Métais E, Meziane F, Saraee M, Sugumaran V, Valtchev P, eds. Natural Language Processing and Information Systems: 29th International Conference on Applications of Natural Language to Information Systems; 25-27 de junio de 2024, Turín, Italia. Springer-Verlag, Berlín, Heidelberg, p. 439-54. https://doi.org/10.1007/978-3-031-70239-6
[10] Trevennec C, Pompidor P, Bououda S, Rabatel J, Roche M. MUST-AI: Multisource Surveillance Tool – Avian Influenza. Procedia Comput. Sci. 2024:246;3034‑43. https://doi.org/10.1016/j.procs.2024.09.718
[11] Roche M. Data sheets highlighting characteristics of PADI-web. París (Francia): Agritrop CIRAD; 2025. Disponible en: https://agritrop.cirad.fr/611480/ (consultado el 23 de mayo de 2025).
[12] Dupuy C, Locquet C, Brard C, Dommergues L, Faure E, Gache K, et al. The French National Animal Health Surveillance Platform: an innovative, cross-sector collaboration to improve surveillance system efficiency in France and a tangible example of the One Health approach. Front. Vet. Sci. 2024:11. https://doi.org/10.3389/fvets.2024.1249925








