Comment transformer des données non structurées en outils pour renforer les systèmes de santé?
L’épidémiologiste vétérinaire Amélie Desvars-Larrive partage les enseignements de deux projets innovants qui transforment les données sanitaires animales en informations puissantes et exploitables, démontrant ainsi comment les données peuvent guider des actions efficaces dans le cadre de la démarche « Une seule santé ».
Les données sanitaires animales désignent toute information liée à la santé des animaux. Elles comprennent les dossiers cliniques, les profils des agents pathogènes, les résultats de laboratoire et les données épidémiologiques. Les données sanitaires animales jouent un rôle essentiel dans la surveillance des maladies animales, le suivi des menaces zoonotiques, la compréhension des schémas de maladies, la protection de la sécurité alimentaire, le soutien à la santé publique et l’orientation des décisions politiques.
Une approche « Une seule santé » réussie repose sur un partage, une intégration, une analyse et une interprétation fluides des données entre les secteurs. Cependant, les données sanitaires animales sont souvent non structurées et dispersées entre différentes agences, plateformes et publications. De plus, elles se présentent sous divers formats et niveaux de granularité, ce qui complique leur intégration et leur analyse. Ces lacunes peuvent entraîner des décisions mal informées, des politiques inefficaces et une mauvaise allocation des ressources.
La pandémie de COVID-19 a mis en évidence ces défis. Alors que les données sur les cas humains étaient rapidement disponibles dans des formats conviviaux sur plusieurs canaux, et malgré les appels mondiaux à une approche « Une seule santé » pour lutter contre la pandémie — ainsi que le premier cas de SARS-CoV-2 rapporté chez des animaux dès février 2020 — les données structurées et facilement exploitables sur le SARS-CoV-2 chez les animaux ont accusé du retard. Cela a révélé une faille critique dans les systèmes de surveillance.
Ce problème dépasse les cadres mondiaux. Lors d’un projet visant à modéliser les interactions zoonotiques naturelles en Autriche, mon équipe a rencontré un obstacle majeur : l’absence d’une base de données nationale complète recensant les sources et agents zoonotiques. Une telle lacune — qui n’est pas propre à l’Autriche — limite notre capacité à comprendre pleinement les systèmes zoonotiques multi-sources et multi-agents.
Au cours des cinq dernières années, notre groupe de recherche a relevé ces défis à travers deux projets axés sur les données, transformant des données sanitaires animales cloisonnées et non structurées en ressources FAIR (Faciles à trouver, Accessibles, Interopérables et Réutilisables) :
- SARS-ANI – Ce projet a compilé le premier jeu de données mondial standardisé des événements SARS-CoV-2 chez les animaux, mettant en lumière des lacunes dans les rapports, notamment une sous-déclaration auprès de l’Organisation mondiale de la santé animale (OMSA), des retards dans la diffusion des cas, et un biais de déclaration en faveur des animaux de compagnie.
- Austrian Zoonotic Web – Ce projet a transformé 47 années (1975–2022) d’enregistrements dispersés en un jeu de données structuré et FAIR sur les interactions zoonotiques en Autriche. Les résultats ont révélé que les espèces domestiques, de gibier et synanthropes représentent le risque le plus élevé de transmission zoonotique en Autriche, et ont mis en évidence l’émergence d’au moins huit agents zoonotiques dans le pays durant cette période.
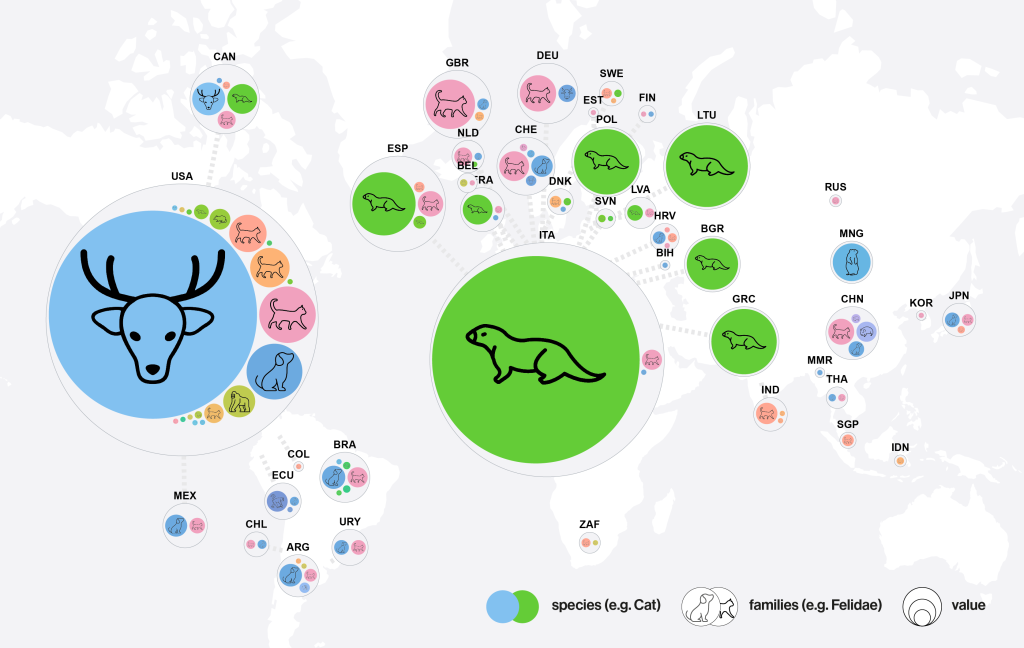
Capture d’écran du tableau de bord SARS-ANI : répartition des événements signalés de SARS-CoV-2 chez les animaux.
Nous considérons comme un « événement » le cas d’un seul animal ou plusieurs cas d’animaux épidémiologiquement liés, identifiés par la présence d’ARN viral et/ou d’anticorps.
La taille du cercle est proportionnelle au nombre d’événements ; les couleurs des cercles correspondent aux espèces animales ; les symboles animaux représentent les familles taxonomiques.
Sources des données : WAHIS et ProMED-mail de la Société internationale pour les maladies infectieuses.
Données extraites de Github le 22 janvier 2025. Image par Amélie Desvars-Larrive, CC BY IGO 3.0
Les deux projets se sont appuyés sur des informations disponibles publiquement. Le jeu de données SARS-ANI consolide les données sur les événements SARS-CoV-2 chez les animaux issues du Programme de surveillance des maladies émergentes (ProMED-mail) et du Système mondial d’information zoosanitaire (WAHIS) de l’Organisation Mondiale pour la Santé Animale (OMSA), de 2020 à aujourd’hui. Nous l’avons ensuite enrichi avec des données issues de prépublications et d’articles évalués par des pairs.
Pour le projet Austrian Zoonotic Web, nous avons effectué une recherche systématique de la littérature scientifique et des rapports nationaux de surveillance afin de collecter des données épidémiologiques sur les interactions zoonotiques signalées en Autriche.
Cependant, l’extraction et la structuration manuelles des données ont été une tâche laborieuse ! Bien que des méthodes automatisées telles que le web scraping et l’apprentissage automatique offrent un grand potentiel, la supervision humaine et l’expertise restent indispensables pour valider des données sanitaires complexes, relier des événements épidémiologiques ou résoudre des incohérences. Pour optimiser ce processus, nous avons utilisé Excel pour la compilation des données et l’environnement logiciel libre R pour un nettoyage systématique, traitant les doublons, les erreurs typographiques et les valeurs manquantes. Des étudiants vétérinaires de la Vetmeduni Vienna ont joué un rôle clé en mobilisant leurs connaissances spécialisées afin d’assurer une extraction et une validation de données de haute qualité.
Une identification précise des espèces — qu’il s’agisse d’animaux, de vecteurs ou d’agents infectieux — est une condition préalable pour éviter les ambiguïtés, garantir l’interopérabilité des données et générer des informations exploitables. L’un de nos plus grands défis ? Les incohérences taxonomiques. De nombreux noms d’espèces étaient obsolètes, incorrects ou incomplets selon les sources. Pour y remédier, nous avons validé et standardisé la nomenclature en utilisant la base de données taxonomique NCBI et le package R taxize.
Pour maximiser l’impact, nous avons suivi les principes directeurs FAIR pour la gestion et la gouvernance des données, ce qui signifie que nos jeux de données étaient :
- Findable (Facile à trouver) – Les données se voyaient attribuer des identifiants d’objets numériques (DOI) pour une identification et une localisation aisées ;
- Accessible – Les jeux de données statiques étaient stockés dans des dépôts ouverts et fiables comme Figshare et Zenodo, garantissant la conservation des données et un accès facile ;
- Interoperable (Interopérable) – Des formats communs comme le .csv et une terminologie standardisée assuraient une utilisation intersectorielle et interdisciplinaire ;
- Reusable (Réutilisable) – Une licence claire (nous avons opté pour la licence CC BY 4.0) et une documentation détaillée soutenaient la réutilisation des données.
Nous ne nous sommes pas arrêtés au simple partage des données. Nous avons fourni des codebooks (détaillant tous les champs et codes utilisés), des scripts R documentés pour explorer les données, reproduire nos résultats et offrir un point de départ pour des analyses complémentaires, ainsi que des fichiers README contenant des instructions pas à pas pour débuter avec R. Cela a rendu les données non seulement accessibles, mais aussi exploitables par d’autres, garantissant transparence et reproductibilité. Nous avons également partagé les sources d’information via des liens intégrés dans le jeu de données ou sous forme de documents dans un dossier séparé.
Pour SARS-ANI, nous sommes allés plus loin en utilisant la plateforme plus flexible GitHub pour des mises à jour régulières — initialement hebdomadaires, maintenant mensuelles — garantissant aux utilisateurs l’accès aux informations les plus récentes.
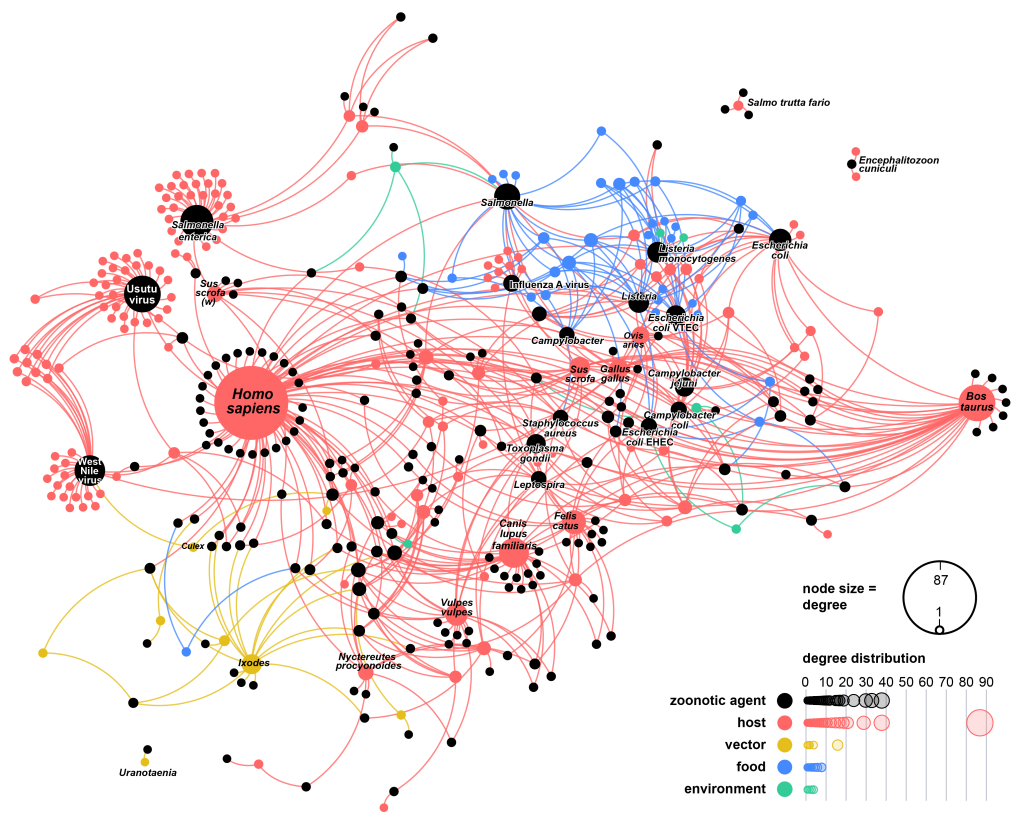
Représentation en réseau des interactions zoonotiques naturelles (« réseau zoonotique ») en Autriche, 1975-2022.
Chaque nœud du réseau (cercle) représente un acteur du réseau zoonotique, avec un ensemble de nœuds (en noir)
représentant les agents zoonotiques et un second ensemble représentant les sources zoonotiques appartenant à différentes catégories :
hôtes vertébrés (rouge), vecteurs arthropodes (jaune), denrées alimentaires (bleu) et environnement (vert).
Un lien entre une source zoonotique et un agent indique que l’agent a été détecté directement ou indirectement dans la source.
Image par Amélie Desvars-Larrive, CC BY 4.0, via Springer Nature
Visualiser les données ne se résume pas à présenter des chiffres ou des graphiques — il s’agit de raconter une histoire. Pour rendre les données complexes sur la santé animale plus accessibles et engageantes, nous avons collaboré avec un expert en visualisation du Complexity Science Hub.
SARS-ANI VIS fut le premier tableau de bord mondial suivant les événements SARS-CoV-2 chez les animaux. Pour le Zoonotic Web, nous avons développé des visuels interactifs, notamment un article en scrollytelling et un tableau de bord interactif. Ces outils ont non seulement amélioré la communication auprès des publics scientifiques, politiques et grand public, mais ont aussi favorisé un engagement accru avec les données sur la santé animale, l’épidémiologie des zoonoses et le concept « Une seule santé ».
La surveillance « Une seule santé » n’est efficace que dans la mesure où les données sur lesquelles elle s’appuie le sont. Sans données équitables, opportunes, de haute qualité et FAIR, notre capacité à prédire, prévenir et répondre aux menaces sanitaires à l’interface homme-animal-environnement reste limitée. C’est pourquoi je suis convaincue que nous devons tous :
- Promouvoir l’accès libre et permanent aux données – Utiliser des dépôts en accès libre ;
- Améliorer les normes de données/métadonnées – Faciliter la découverte et la réutilisation des données ;
- Adopter une approche “human-in-the-loop” – Combiner outils computationnels et validation experte pour garantir la fiabilité et la précision des données ;
- Investir dans la culture des données – Former les professionnels à la gestion et à l’analyse des données, notamment dans le personnel vétérinaire ;
- Reconnaître les contributions – Valoriser les sources et les équipes pour leurs efforts ;
- Renforcer la communication – Créer des visualisations conviviales et attractives pour toucher des publics variés.
En relevant ces défis collectivement, nous pourrons renforcer les données sur la santé animale, améliorant ainsi la surveillance intégrée « Une seule santé » et les mécanismes d’alerte précoce, pour un futur où les données guideront des décisions sanitaires plus intelligentes, rapides et efficaces.
Agradecimientos
Quisiera expresar mi más sincero agradecimiento a Eliza Muto, Anja Böck, Liuhuaying Yang y Gavrila Amadea Puspitarani por sus valiosos comentarios sobre el texto.
The Animal Echo vise à promouvoir la compréhension individuelle et collective de la santé et du bien-être des animaux. Nous vous présentons des idées et des opinions d’experts en matière de santé et de bien-être des animaux dans le monde entier. Les opinions exprimées dans The Animal Echo sont celles de(s) auteur(s) et ne reflètent pas nécessairement la position officielle de l’OMSA.







