Résumé
Depuis le début des années 2000, plusieurs systèmes de surveillance fondée sur les événements (EBS) ont été créés pour collecter les informations sur les épidémies publiées dans les médias en ligne (par exemple, Promed-mail depuis 1994, HealthMap depuis 2006, PADI-web depuis 2016, EIOS depuis 2017) pour une série de maladies et de syndromes chez les humains et les animaux. Ces EBS constituent des systèmes d’alerte sur les événements sanitaires, permettant aux épidémiologistes de mener une veille sanitaire précoce, y compris dans les zones faiblement couvertes par les systèmes de surveillance officiels. Une partie de ces systèmes EBS, comme PADI-web, intègre des méthodes d’intelligence artificielle (IA) et de traitement automatique du langage naturel (TALN) pour améliorer les processus de classification automatique de documents ou pour identifier des événements épidémiologiques dans les articles. Cet article donnera des exemples de l’utilisation quotidienne des systèmes EBS en France.

Figure 1. Processus de traitements de PADI-web
L’intelligence épidémique, est une discipline d’importance croissante pour l’identification globale et précoce des maladies animales infectieuses et émergentes, susceptibles de mettre en péril la bonne santé d’un cheptel national, la santé publique (maladies zoonotiques) et la conservation des espèces (races à faibles effectifs, espèces sauvages). Elle a pour objectif d’identifier précocement, d’analyser et de suivre des signaux relatifs aux dangers sanitaires menaçant la santé animale. Elle repose sur la surveillance en continu des signaux de nature variée issus de sources officielles et non officielles (médias, réseaux sociaux, etc.).
L’une des missions de l’Organisation mondiale de la santé animale (OMSA) est d’améliorer la détection précoce et la diffusion d’informations sur l’apparition de maladies animales au niveau mondial. Les membres de l’OMSA mettent à disposition les informations sanitaires officielles relatives aux maladies animales réglementées dans la base de données WAHIS (World Animal Health Information System), sous forme structurée et standardisée. Les experts en intelligence épidémique peuvent analyser ces données selon une routine prédéfinie, qui permet de produire des indicateurs épidémiologiques et d’effectuer des bilans périodiques de situation. Cependant, les données officielles peuvent subir des biais ou des délais de notification liés à de multiples facteurs [1].
Une nouvelle approche : des événements sanitaires extraits sur le Web
Depuis les années 2000, une nouvelle génération de systèmes de surveillance de la santé complète le système existant en identifiant les événements sanitaires extraits d’Internet et d’autres médias électroniques. C’est la surveillance fondée sur les événements, dite EBS [2]. De nombreuses interfaces ont émergé dans le secteur de la santé publique, avec un volet dédié à la santé animale, telles que Promed en 1994, HealthMap en 2006, PADI-web en 2016 et EIOS en 2017. Elles permettent d’identifier, d’extraire, de classer et de visualiser les événements sanitaires issus d’informations textuelles et non structurées.
Une partie de ces systèmes EBS comme PADI-web (Platform for Automated extraction of animal Disease Information from the Web) [3,4] intègre des méthodes d’intelligence artificielle (IA) [5] et de traitement automatique du langage naturel (TALN) pour améliorer les processus de classification automatique de dépêches (voir étapes 3 et 4 de la Figure 1) ou pour identifier des informations épidémiologiques dans les articles (voir étape 5 de la Figure 1). Des interfaces dédiées à la recherche d’informations (Figure 2) et à la visualisation des événements épidémiologiques sont proposées (Figure 3).
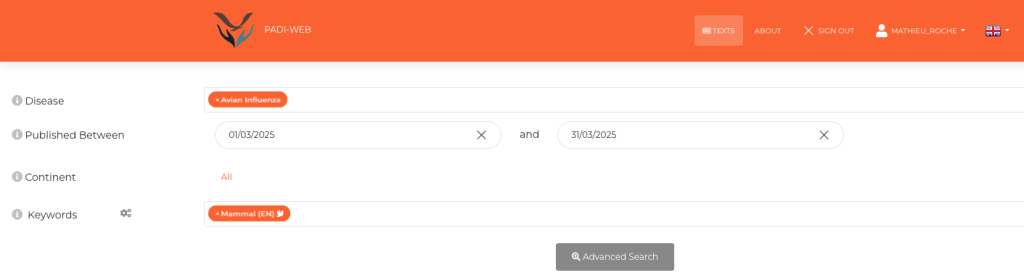
Figure 2. Interface de recherche de PADI-web
Pour mettre en œuvre et adapter les approches de l’IA au service de la veille en santé animale, PADI-web utilise des données manuellement annotées avec des experts. Les données brutes qui sont collectées et annotées pour la santé animale représentent un matériau précieux qui permet d’apprendre des modèles spécifiques en utilisant par exemple des méthodes d’apprentissage automatique. Par ailleurs, les approches de l’IA fondées sur les modèles de langue généralistes (par exemple, BERT – Bidirectional Encoder Representations from Transformers [6]) ou plus spécialisés (par exemple, BioBERT pour le domaine biomédical [7], AgriBERT pour l’agriculture [8]) permettent d’adapter les modèles à des domaines dédiés et aux tâches à réaliser. BERT est un modèle de langue fondé sur une architecture spécifique avec un mécanisme d’attention qui permet de comprendre les relations entre les mots de la phrase.
Dans le cadre du projet MOOD (MOnitoring Outbreaks for Disease surveillance in a data science context) coordonné par Elena Arsevska, le CIRAD (Centre de coopération internationale en recherche agronomique pour le développement) et l’INRAE (Institut national de recherche pour l’agriculture, l’alimentation et l’environnement) ont mené des travaux pluridisciplinaires pour mettre en place et intégrer des modèles d’IA dans les outils de veille épidémiologique. Pour construire des modèles d’apprentissage automatique traditionnels (Support Vector Machine, Random Forest, etc.) et/ou pour ajuster des modèles de langues, des corpus (ensemble de dépêches) dédiés ont été constitués à travers des séances d’annotation de données avec des experts en santé animale et l’organisation d’« hackathons » (travail collectif dédié à des tâches spécifiques). Ces séances d’annotation peuvent s’appuyer sur des méthodes spécifiques comme la méthode Delphi (plusieurs tours d’annotation pour converger vers un consensus) et des calculs d’accord entre annotateurs pour consolider des guides d’annotation et améliorer la qualité des données produites qui sont utilisées par les algorithmes d’IA.
Les corpus utilisés sont annotés à l’échelle de l’article en déterminant si une dépêche est pertinente ou non. Une dépêche est considérée comme pertinente si le texte décrit un foyer épidémique nouveau, suspect ou inconnu. Pour construire un modèle d’extraction d’informations à partir des textes pertinents, les informations épidémiologiques (par exemple, les maladies, les hôtes, les lieux, les dates, etc.) doivent également être extraites dans le contenu textuel. De manière générale, les modèles d’IA appris et adaptés en mettant en place des réglages fins à partir de ces données annotées sont aujourd’hui intégrés au système PADI-web dans un cadre opérationnel.
Ces approches de l’IA permettent de déterminer automatiquement (i) si les documents automatiquement collectés sur le web par un processus d’analyse et d’indexation sont pertinents, (ii) quels sont les thèmes qu’ils véhiculent, et (iii) quels sont les informations épidémiologiques (maladies, lieux des foyers, hôtes, symptômes, nombre de cas, etc.) dans les textes. Cette intégration prend en compte les aspects qualitatifs des résultats mais également la frugalité des algorithmes mobilisés, ce qui constitue un autre enjeu crucial des méthodes d’IA actuellement développées.
Aujourd’hui, de multiples autres enjeux sont étudiés, par exemple pour identifier des signaux faibles, des événements identiques ou nouveaux, etc. Dans ce cadre, les modèles de langue et les grands modèles de langue (techniques de modèles génératifs fondées sur ChatGPT et autres) peuvent se révéler particulièrement efficaces, comme des travaux en collaboration entre des chercheurs de Strathmore University (Kenya) et du CIRAD le montrent [9].
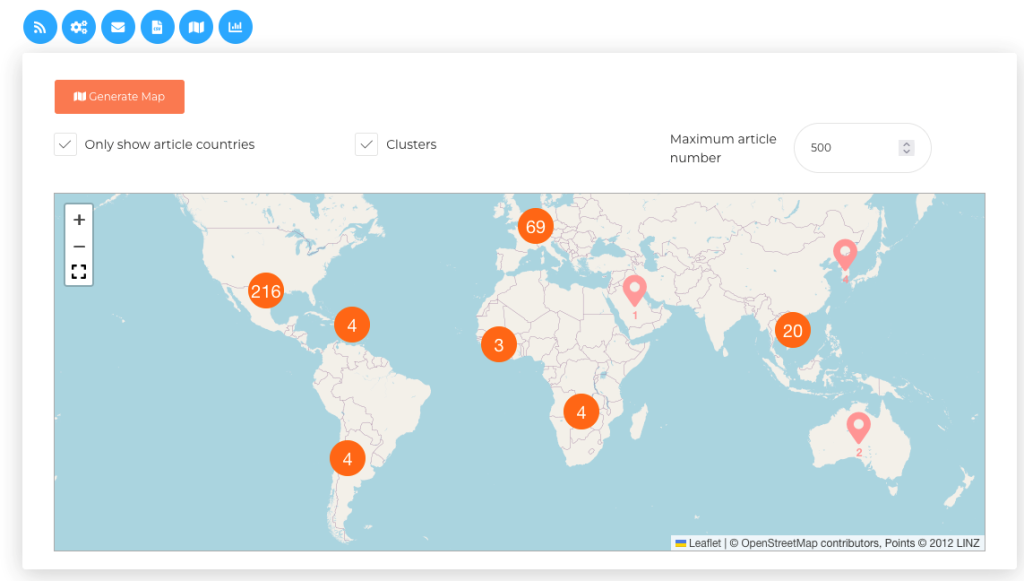
Figure 3. Interface de visualisation spatiale d’événements épidémiologiques de PADI-web
Des travaux menés entre le CIRAD et l’INRAE sont également dédiés à la veille syndromique en santé animale qui constitue un autre défi permettant d’identifier de nouvelles maladies potentielles sur de nouveaux terrains ou avec des hôtes nouveaux. Dans ce contexte, une analyse rétrospective de maladie émergente telle que la détection du virus de l’influenza aviaire de haute pathogénicité (IAHP) chez des espèces hôtes inhabituelles a été réalisée. Sept cas d’étude d’IAHP chez des mammifères ont été identifiés dans la base de données WAHIS, pour lesquels les articles collectés par PADI-web ont été validés manuellement [10]. Plusieurs stratégies de classification des lieux ont été évaluées, de manière à privilégier la sensibilité ou la spécificité du système de surveillance.
En parallèle de ces travaux de recherche menées dans le cadre du projet MOOD, des séances de formation avec les utilisateurs finaux ainsi que des fiches pédagogiques [11] ont été produites pour faciliter l’utilisation de cet outil.
La cellule d’intelligence épidémique de la plateforme française d’épidémiosurveillance des maladies animales (plateforme ESA) [12] utilise au quotidien de tels systèmes EBS. Aussi, un des enjeux aujourd’hui consiste à fusionner ces différentes informations épidémiologiques issues de sources variées. Dans ce contexte, un nouvel outil appelé MUST (Multi-Source Surveillance Tool) a été développé pour établir la correspondance entre diverses sources de données sanitaires [10]. Le premier volet de MUST est consacré à la surveillance des foyers d’influenza aviaire de haute pathogénicité chez les mammifères (IAHP-M).
L’outil collecte, filtre et cartographie les événements d’IAHP-M issus des notifications officielles extraites de la base de données WAHIS, les données officielles issues de bases de données disponibles en ligne gérées par les autorités sanitaires (USDA pour les États-Unis d’Amérique et APHA pour le Royaume-Uni) et les événements issus de différents EBS (Promed-mail et PADI-web). L’outil est actuellement en phase de test pour évaluer les différentes stratégies de fusions selon des critères spatiaux, temporels et les informations sur les hôtes impliqués dans les évènements épidémiologiques. Cette expertise permettra de proposer des démarches à appliquer pour les futurs utilisateurs de MUST.
Les approches d’IA peuvent se révéler particulièrement efficaces pour traiter les problématiques de veille en santé animale. Cependant, ces travaux nécessitent des connaissances expertes pour annoter, valider les résultats des algorithmes, adapter les paramètres et rendre les outils opérationnels ce qui nécessite de mettre en place des recherches pluridisciplinaires.
Copyright de l’image principale : akinbostanci
Références
[1] Lin SY, Beltran-Alcrudo D, Awada L, Hamilton-West C, Lavarello Schettini A, Cáceres P, et al. Analysing WAHIS Animal Health Immediate Notifications to Understand Global Reporting Trends and Measure Early Warning Capacities (2005–2021). Transbound. Emerg. Dis. 2023. https://doi.org/10.1155/2023/6666672
[2] Paquet C, Coulombier D, Kaiser R, Ciotti M. Epidemic intelligence: a new framework for strengthening disease surveillance in Europe. Euro Surveill. 2026:11(12);5‑6. https://doi.org/10.2807/esm.11.12.00665-en
[3] Arsevska E, Valentin S, Rabatel J, de Goër de Hervé J, Falala S, Lancelot R, et al. Web monitoring of emerging animal infectious diseases integrated in the French Animal Health Epidemic Intelligence System. PLoS One. 2018:13(8);25. https://doi.org/10.1371/journal.pone.0199960
[4] Valentin S, Arsevska E, Rabatel J, Falala S, Mercier A, Lancelot R, et al. PADI-web 3.0: A new framework for extracting and disseminating fine-grained information from the news for animal disease surveillance. One Health. 2021:13. https://doi.org/10.1016/j.onehlt.2021.100357
[5] Sobkowich KE. Demystifying artificial intelligence for veterinary professionals: practical applications and future potential. Am. J. Vet. Res. 2025:86. https://doi.org/10.2460/ajvr.24.09.0275
[6] Devlin J, Chang MW, Lee K, Toutanova K. BERT: Pre-training of Deep Bidirectional Transformers for Language Understanding. Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. 2019:1;4171-86. https://doi.org/10.18653/v1/N19-1423
[7] Lee J, Yoon W, Kim S, Kim D, Kim S, Ho So C, et al. BioBERT: a pre-trained biomedical language representation model for biomedical text. Bioinformatics. 2020:36(4);1234-40. https://doi.org/10.1093/bioinformatics/btz682
[8] Rezayi S, Liu Z, Wu Z, Dhakal C, Ge B, Zhen C, et al. AgriBERT: Knowledge-Infused Agricultural Language Models for Matching Food and Nutrition. Proceedings of the Thirty-First International Joint Conference on Artificial Intelligence – AI for Good. 2022;5150-56. https://doi.org/10.24963/ijcai.2022/715
[9] Menya E, Roche M, Interdonato R, Owuor D. EpidGPT: A combined strategy to discriminate between redundant and new information for epidemiological surveillance systems. In: Métais E, Meziane F, Saraee M, Sugumaran V, Valtchev P, eds. Natural Language Processing and Information Systems: 29th International Conference on Applications of Natural Language to Information Systems; 2024 June 25-27, Turin, Italie. Springer-Verlag, Berlin, Heidelberg, p. 439-54. https://doi.org/10.1007/978-3-031-70239-6
[10] Trevennec C, Pompidor P, Bououda S, Rabatel J, Roche M. MUST-AI: Multisource Surveillance Tool – Avian Influenza. Procedia Comput. Sci. 2024:246;3034‑43. https://doi.org/10.1016/j.procs.2024.09.718
[11] Roche M. Data sheets highlighting characteristics of PADI-web. Paris (France): Agritrop CIRAD; 2025. Disponible en ligne : https://agritrop.cirad.fr/611480/ (consulté le 23 mai 2025).
[12] Dupuy C, Locquet C, Brard C, Dommergues L, Faure E, Gache K, et al. The French National Animal Health Surveillance Platform: an innovative, cross-sector collaboration to improve surveillance system efficiency in France and a tangible example of the One Health approach. Front. Vet. Sci. 2024:11. https://doi.org/10.3389/fvets.2024.1249925








